„You never walk alone“ is a beautiful catch phrase equally relevant for social communities as well as the subcellular organization at the molecular level. Very much like best friends also proteins, cellular micromachines, come in close contact and team up for a shared mission. For example, most processes vital to life such as replication of DNA, energy production by mitochondria, movement of muscles, or the release of hormones like insulin rely on the interactions of different proteins.
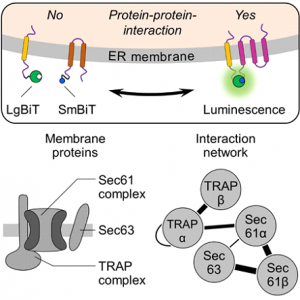 Fascinated by the output that originates from the teamwork of proteins, our lab established a method to track and measure individual protein-protein interactions in living cells. We focused on the protein translocase of the endoplasmic reticulum, a multimeric protein complex that is necessary for the transport of hundreds of other proteins including the aforementioned hormone insulin. While the interactions between subunits of the protein translocase have been addressed by biochemical co-purification methods or structural X-ray crystallography before, we managed to detect and modulate those interactions for the first time in living cells. In addition, we identified interactions of other membrane proteins of the endoplasmic reticulum that have not been shown in mammalian cells before.
Fascinated by the output that originates from the teamwork of proteins, our lab established a method to track and measure individual protein-protein interactions in living cells. We focused on the protein translocase of the endoplasmic reticulum, a multimeric protein complex that is necessary for the transport of hundreds of other proteins including the aforementioned hormone insulin. While the interactions between subunits of the protein translocase have been addressed by biochemical co-purification methods or structural X-ray crystallography before, we managed to detect and modulate those interactions for the first time in living cells. In addition, we identified interactions of other membrane proteins of the endoplasmic reticulum that have not been shown in mammalian cells before.
The principle of the assay relies on a molecular lightbulb called luciferase. The clue, this lightbulb can be disassembled into the bulb and the glow filament that are individually attached to proteins of interest using standardized molecular biological methods. If those fusion proteins interact, the bulb and glow filament are brought together and emit light that can be detected by a camera. Thus, like all celebrated movie stars our subunits of the protein translocase were just waiting for the famous words “Lights. Camera. (Inter)action.” of the movie director and first author Mark Sicking. Using the expertise of the team’s chemist, Prof. Dr. Martin Jung, different types of glow filaments (biomimetic peptides) were designed that allowed us to determine the presence and localization of the bulb part within cells. Overall, this work laid the cornerstone for further studies that will address the dynamic behavior of protein-protein interactions in cellular systems. Further information about our team can be found at https://www.molmed-saarland.eu/team-lang/. (Text/Foto by Sven Lang; from left to right: Sven Lang, Mark Sicking, Martin Jung)
Original publication
Sicking M, Jung M, Lang S (2021) Lights, Camera, Interaction: Studying Protein–Protein Interactions of the ER Protein Translocase in Living Cells. Int. J. Mol. Sci. 22, 10358; https://doi.org/10.3390/ijms221910358
“You never walk alone” ist eine schöne Redewendung, die sowohl für soziale Gemeinschaften als auch für die subzelluläre Organisation auf molekularer Ebene gilt. Ähnlich wie beste Freunde stehen auch Proteine, zelluläre Mikromaschinen, in engem Kontakt und arbeiten zusammen auf ein gemeinsames Ziel hin. So sind beispielsweise die meisten lebenswichtigen Prozesse wie die Replikation der DNA, die Energieerzeugung durch Mitochondrien, die Kontraktion von Muskeln oder auch die Freisetzung von Hormonen wie Insulin auf das Zusammenspiel unterschiedlicher Proteine angewiesen.
Fasziniert von der Leistung, die aus dieser Kooperation von Proteinen entsteht, hat unser Labor eine Methode etabliert, mit der sich einzelne Protein-Protein-Interaktionen in lebenden Zellen verfolgen und messen lassen. Wir konzentrierten uns auf die Proteintranslokase des endoplasmatischen Retikulums, ein multimerer Proteinkomplex, der für den Transport von Hunderten anderer Proteine, darunter das bereits erwähnte Hormon Insulin, notwendig ist. Während die Interaktionen zwischen den Untereinheiten der Proteintranslokase bereits durch biochemische Aufreinigungen oder strukturelle Röntgenkristallographie untersucht wurden, gelang es uns, zum ersten Mal, diese Wechselwirkungen auch in lebenden Zellen nachzuweisen und zu modulieren. Darüber hinaus haben wir Interaktionen anderer Membranproteine des endoplasmatischen Retikulums identifiziert, die bisher nicht in Säugetierzellen nachgewiesen wurden.
Das Prinzip der Methode selbst beruht auf einer molekularen Glühbirne namens Luziferase. Diese Glühbirne kann in den Glaskolben und die Glühwendel separiert werden, um jeweils eines der Bauteile mittels molekularbiologischer Verfahren an ein Protein von Interesse anzuhängen. Interagieren diese Fusionsproteine miteinander, werden Glaskolben und Glühwendel zusammengebracht und senden Licht aus, das von einer Kamera erfasst werden kann. Wie alle gefeierten Filmstars warteten auch unsere Untereinheiten der Proteintranslokase nur auf die berühmte Phrase “Licht. Kamera. (Inter-)Aktion.” des Filmregisseurs und Erstautors Mark Sicking. Mithilfe des Chemikers Prof. Dr. Martin Jung wurden diverse Varianten der Glühwendel (biomimetische Peptide) generiert. Diese ermöglichten es uns das Vorhandensein und die Lokalisierung der Glühbirne innerhalb von Zellen zu bestimmen. Insgesamt legte diese Arbeit den Grundstein für weitere Studien, die sich mit dem dynamischen Verhalten von Protein-Protein-Interaktionen in zellulären Systemen befassen werden. Weitere Informationen über unser Team finden sich unter https://www.molmed-saarland.eu/team-lang/. (Text/Foto von Sven Lang; von links nach rechts: Sven Lang, Mark Sicking, Martin Jung)
Originalpublikation
Sicking M, Jung M, Lang S (2021) Lights, Camera, Interaction: Studying Protein–Protein Interactions of the ER Protein Translocase in Living Cells. Int. J. Mol. Sci. 22, 10358; https://doi.org/10.3390/ijms221910358

